KI identifiziert Mutationen. Karl Landsteiner Privatuniversität für Gesundheitswissenschaften setzt erneut Standards durch Machine Learning-Methoden
Krems (Österreich), 16. Mai 2024: Machine Learning (ML)-Methoden können Mutationen in Gliomen – primären Hirntumoren – rasch und akkurat diagnostizieren. Das zeigt eine aktuelle Studie der Karl Landsteiner Privatuniversität für Gesundheitswissenschaften (KL Krems). In dieser wurden Daten von physio-metabolischen Magnetresonanz-Bildern zur Identifikation von Mutationen eines Stoffwechselgens mittels ML ausgewertet. Mutationen dieses Gens beeinflussen den Krankheitsverlauf maßgeblich, und eine frühe Diagnose ist für die Behandlung wichtig. Die Studie zeigt auch, dass dem klinischen Routineeinsatz der Methode aktuell jedoch noch uneinheitliche Standards bei der Gewinnung physio-metabolischer Magnetresonanz-Bilder entgegenstehen.
Gliome sind die häufigsten Primärtumore des Gehirns. Trotz noch immer schlechter Prognose können personalisierte Therapien den Behandlungserfolg aber bereits maßgeblich verbessern. Doch der Einsatz solcher modernen Therapien beruht auf individuellen Tumordaten, die bei Gliomen aufgrund ihrer Lokalisation im Gehirn nicht leicht verfügbar sind. Bildgebende Verfahren wie die Magnetresonanztomographie (MRT) können solche Daten liefern, ihre Auswertung ist jedoch komplex, anspruchsvoll und zeitaufwendig. Das Zentralinstitut für medizinische Radiologie-Diagnostik des Universitätsklinikums St. Pölten, einem Lehr- und Forschungsstandort der KL Krems, entwickelt daher seit Jahren Methoden des Machine und Deep Learning, um solche Auswertungen zu automatisieren und so in den Routinebetrieb von Kliniken integrieren zu können. Nun gelang dort ein weiterer Durchbruch.
POSITIVE MUTATION
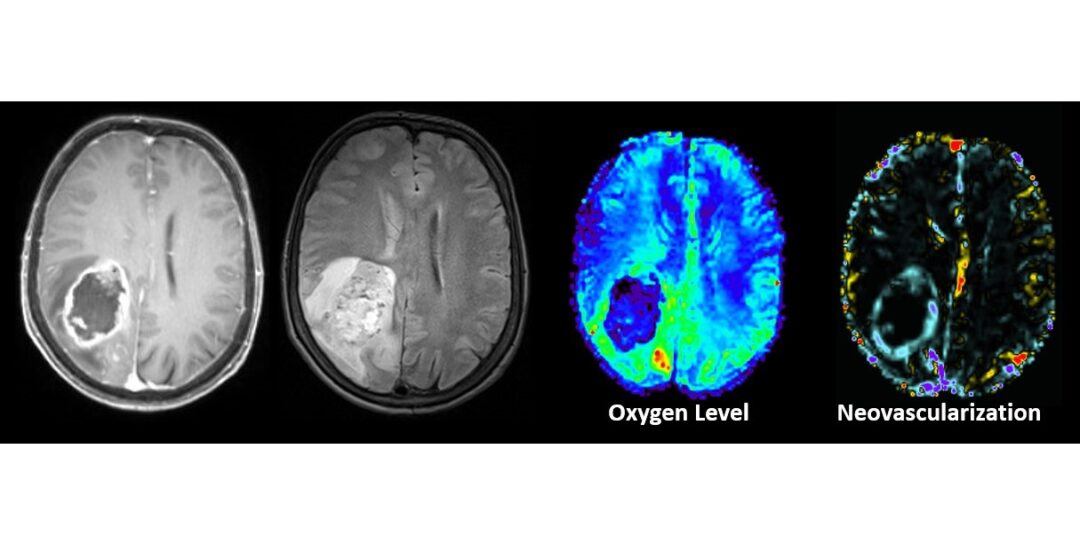
„Tatsächlich haben Patientinnen und Patienten, deren Gliomzellen eine mutierte Form des Gens für Isocitratdehydrogenase (IDH) tragen,“ erläutert Prof. Andreas Stadlbauer, Medizinphysiker am Zentralinstitut, „bessere klinische Aussichten als jene, bei denen die Wildtypform vorliegt. Das bedeutet: Je früher wir über diesen Mutationsstatus Bescheid wissen, desto optimaler können wir die Behandlung individualisieren.“ Dabei helfen Unterschiede im Energiestoffwechsel von mutierten bzw. Wildtyp-Tumoren. Diese können –dank früheren Arbeiten des Teams um Prof. Stadlbauer – auch ohne Gewebeproben mittels sogenannter physio-metabolischer MRT gut erfasst werden. Doch die Auswertung und Beurteilung der Daten sind eine hochkomplexe und zeitraubende Angelegenheit, die schwer in den klinischen Routinebetrieb zu integrieren ist, zumal Ergebnisse aufgrund der schlechten Prognose für Betroffene rasch erforderlich sind.
In der aktuellen Studie hat das Team daher diese Daten mittels ML-Methoden analysiert und ausgewertet, um auf diese Weise rascher ein Ergebnis zu erhalten und entsprechende Therapieschritte einleiten zu können. Aber wie präzise sind die dabei erzielten Ergebnisse? Um dies zu beurteilen, wurde in der Studie zunächst auf Daten von 182 Patientinnen und Patienten des Universitätsklinikums St. Pölten zurückgegriffen, deren MRT-Daten nach einheitlichen Protokollen erhoben wurden.
POSITIVE ERGEBNISSE
„Als wir die Ergebnisse der Beurteilung durch unsere ML-Algorithmen sahen,“ führt Prof. Stadlbauer aus, „waren wir sehr erfreut. Wir erzielten eine Präzision von 91,7 % und eine Genauigkeit von 87,5 % bei der Unterscheidung zwischen Tumoren mit dem Wildtyp-Gen oder der mutierten Form. Wir verglichen diese Werte dann auch mit ML-Auswertungen von klassischen klinischen MRT-Daten und konnten zeigen, dass die Verwendung von physio-metabolischen MRT-Daten als Grundlage eindeutig bessere Ergebnisse erzielte.“
Diese Überlegenheit galt jedoch nur so lang, wie die Analyse an den in St. Pölten – nach einheitlichem Protokoll – erhobenen Daten erfolgte. Das zeigte sich, als die ML-Methode auf externe Daten, also MRT-Daten aus Datenbanken anderer Krankenhäuser, angewendet wurde. In diesem Fall erwies sich die ML-Methode als erfolgreicher, die mit klassischen klinischen MRT-Daten trainiert worden war. „Dass die ML-Auswertung der physio-metabolischen MRT-Daten hier schlechter abschnitt, liegt daran, dass die Technik noch jung und in einer experimentellen Entwicklungsphase ist. Da variieren die Methoden zur Datenerhebung noch von Krankenhaus zu Krankenhaus, was zu Verzerrungen in der ML-Auswertung führt“, so Stadlbauer.
Doch dies Problem ist für den Wissenschafter „nur“ eines der Standardisierung, die mit zunehmendem Einsatz physio-metabolischer MRTs an verschiedenen Krankenhäusern unweigerlich kommen wird. Die Methode selbst – die zeitsparende Auswertung physio-metabolischer MRT-Daten durch ML-Methoden – hat sich großartig bewährt. Sie bietet damit einen hervorragenden Ansatz, um zukünftig präoperativ den IDH-Mutationsstatus von Gliom-Betroffenen zu erheben und Therapieoptionen zu individualisieren.
Links und vollständige Kontaktdetails hier: https://prd.at/newsroom-kunden/fortschritt-fuer-die-personalisierte-therapie-von-hirntumoren/
Originalpublikation: Machine Learning-Based Prediction of Glioma IDH Gene Mutation Status Using Physio-Metabolic MRI of Oxygen Metabolism and Neovascularization (A Bicenter Study). A. Stadlbauer, K. Nikolic, S. Oberndorfer, F. Marhold, T. M. Kinfe, A. Meyer-Bäse, D. Alina Bistrian, O. Schnell & A. Doerfler. Cancers 2024, 16, 1102.
Foto © Andreas Stadlbauer